Nature Biotechnology | 王皓毅研究员合作发现最小 RNA 介导基因编辑工具
 发布时间:2023-07-02
发布时间:2023-07-02
 信息来源:怀柔科学城微信公众号
字体:
大
中
小
信息来源:怀柔科学城微信公众号
字体:
大
中
小
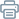 打印
打印
 发布时间:2023-07-02
发布时间:2023-07-02
 信息来源:怀柔科学城微信公众号
信息来源:怀柔科学城微信公众号
- 蛋白;基因;编辑器;编辑工具;基因编辑;核酸酶

基因编辑技术(Genome editing)可谓近些年生命科学领域最重要的发现之一。该技术可实现基因组特定位点的精确修饰,能够在目标基因位点插入、缺失或替换 DNA 序列。因此,基因编辑技术迅速成为生物学研究通用的核心技术,方便了科研探索;更重要的是,该技术还可以直接应用于遗传病的治疗、农业、畜牧业育种改造等方面,成为保障人民生命健康和改善生活质量的重要工具。
当前,最常用的 CRISPR-Cas 基因编辑系统 Cas9 和 Cas12 已经较为成熟;在这些系统中,Cas 蛋白在特定非编码RNA的引导下识别基因组中的目标位点,进而发挥核酸酶靶向剪切 DNA 的功能。然而,这些 Cas 蛋白大都超过 1000 个氨基酸,超出了基因治疗递送常用载体 AAV(Adeno-associated virus)的承载能力,使得递送它们进入目的细胞变得很困难。Cas 蛋白通过融合脱氨酶或逆转录酶可以成为更为精确的碱基编辑和引导编辑工具,但融合蛋白尺寸更大,其有效表达和递送更为困难。
Cas12 核酸酶的祖先 TnpB 由 IS200/IS605 等原核转座子家族编码,并被推测参与转座子的扩张。最新研究进展发现 TnpB 蛋白尺寸更小,且同样能在非编码 RNA(omegaRNA 或 reRNA)引导下切割 DNA,具有和 CRISPR-Cas 系统相似的工作机制。因此,TnpB 有望被开发为新的微型基因编辑工具,解决实际应用中因蛋白过大而递送困难的问题。不仅如此,TnpB 在目前已知的基因组存在中存在超过百万份拷贝,是一个尚未被开发的巨大宝库。筛选具有靶向编辑活性的 TnpB 蛋白,无疑将推进基因编辑工具的改良和应用。
2023 年 6 月 29 日,北京干细胞与再生医学研究院/中国科学院动物研究所王皓毅研究员、项光海博士和中国科学院动物研究所张勇研究员团队合作在 Nature Biotechnology 杂志在线发表题为 Evolutionary mining and functional characterization of TnpB nucleases identify efficient miniature genome editors 研究论文,报道了关于 TnpB 基因编辑系统开发的研究进展。

文章链接:https://www.nature.com/articles/s41587-023-01857-x
研究团队首先对 ISfinder 原核转座子数据库中 IS605 编码的 TnpB 蛋白进行了全面的分析和挖掘,从 64 个候选项中鉴定出 25 种在大肠杆菌中活跃的系统,其中 3 种在人类细胞中具有基因编辑活性。
对功能数据的进一步分析则揭示 TnpB 蛋白相关的 reRNA 骨架与 IS200/IS605 转座子的 RE 序列具有完全重叠的 3’末端;而最优 TAM 序列则与转座子上游的插入位点序列相同。该结果表明在 TnpB 系统中,RNA 介导的编辑器相关的三大要素 —— 核酸酶、gRNA 骨架和 TAM 序列(分别相当于 CRISPR-Cas 系统的 Cas9 蛋白、gRNA和PAM),均可通过生物信息分析准确预测,为大规模筛选高活性 TnpB 核酸酶奠定了基础。这一发现同时也说明 TnpB 作为归巢核酸酶,通过切割 IS605 转座之后的原位点诱导重组修复从而实现转座子的拷贝数扩增。
随后,研究团队对 reRNA 骨架以及影响 TnpB 编辑器活性的多方面因素进行了全面分析,结果显示:
1)reRNA 骨架在 120-300nt 的长度范围内均能够有效发挥功能,而 120-140nt 的 reRNA 骨架活性最强;
2)reRNA 骨架在 3’ 末端的碱基对其功能有重要影响,单一碱基的突变即会显著降低编辑活性;
3)靶向序列的长度在 16-20nt 为最佳;
4)靠近 TAM 端的 12nt 是 TnpB 编辑器的核心序列;
5)来自细菌的、由多拷贝转座子编码的、具有完整蛋白结构域和保守氨基酸的 TnpB 编辑器更倾向于活跃。
基于上述理解,团队建立了建立了适用于 TnpB 编辑器的大规模注释预测和筛选体系。
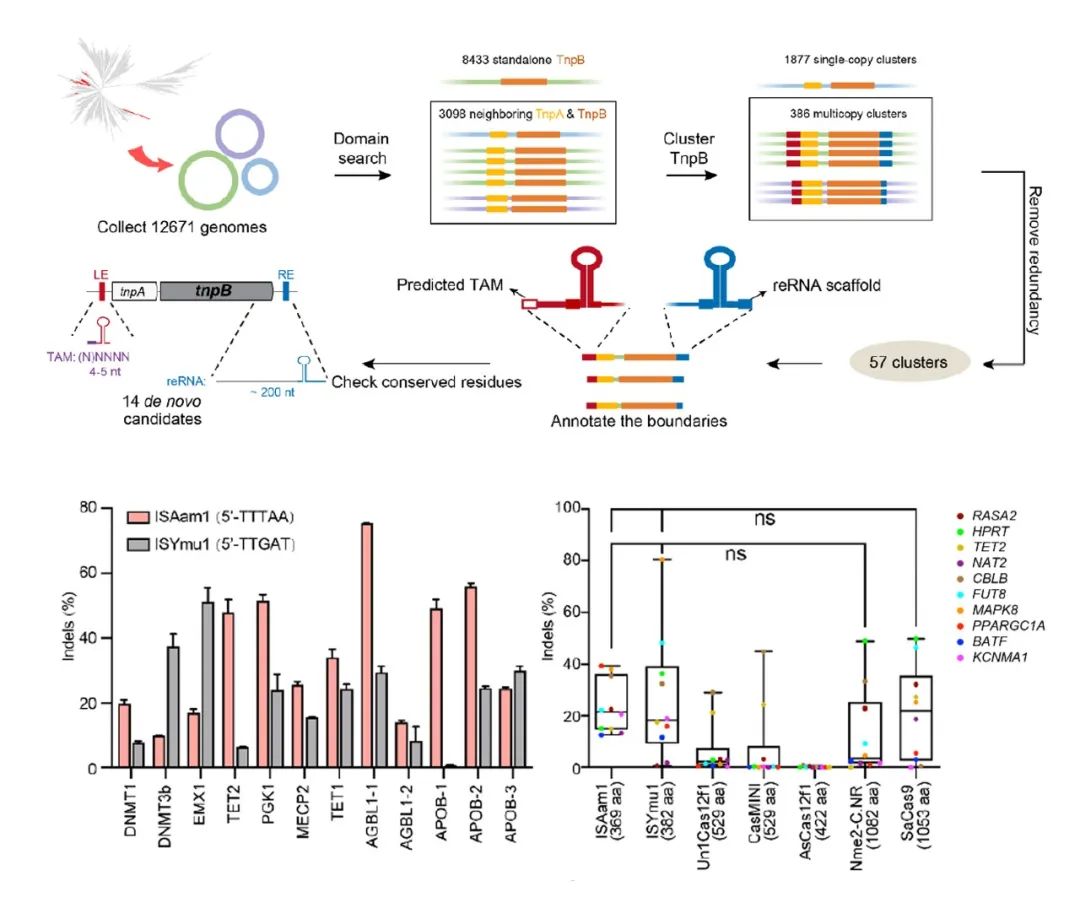
利用该体系,研究者从原核基因组中挖掘到了迄今发现的最小的 RNA 介导的基因编辑工具 ISAam1(369个氨基酸)和 ISYmu1(382个氨基酸)。和其他微型 Cas 蛋白的平行比较发现,ISAam1 和 ISYmu1 的活性与 SaCas9 相当,显著高于数种已报道的 Cas12f 蛋白及其变体。
TnpB 基因编辑器从头筛选路径以及在哺乳动物细胞中基因编辑活性的验证和比较
ISAam1 和 ISYmu1 极小的蛋白尺寸不仅能够适配不同的递送平台,而且在与脱氨酶、逆转录酶等衍生功能模块融合后仍然较小,可以利用 AAV 递送。这将有力推动在体基因治疗的发展,有望为基因缺陷导致的疾病,如 TTR 相关淀粉样变性病、β 地中海贫血及较常见的高胆固醇病等提供更为有效的治疗新方案。
综上,该研究建立了适用于 TnpB 编辑器的大规模筛选体系,进一步证明了 TnpB 在转座子扩张中的功能,并对这一类编辑器进行了系统的功能解析,从而获得了目前最小的微型基因编辑器 ISAam1 和 ISYmu1,这将推动因 Cas 蛋白过大而递送受限的体内基因治疗和细胞治疗的研究和临床应用。
另外值得一提的是,本研究发现的新型基因编辑工具已申请国际专利,有助于我国发展基于基因编辑底层工具的相关产业。更为重要的是,本研究为未来针对基因组数据中数以百万计的 TnpB 位点的高通量挖掘提供了高效通用的平台,后续基于这一平台的进一步挖掘将发现更多、更好的基因编辑工具,服务于全人类的健康与生活。
该研究由北京干细胞与再生医学研究院/中国科学院动物研究所完成。北京干细胞与再生医学研究院/中国科学院动物研究所王皓毅研究员、项光海博士和中国科学动物研究所张勇研究员为文章的共同通讯作者。项光海博士、研究生李源清、孙晶、霍雍元为文章的共同第一作者。
王皓毅研究员长期致力于新型基因编辑工具的开发(Cell Discovery 2019)及 CAR-T 细胞治疗研究(Cancer Cell 2022);张勇研究员则聚焦于转座子等机制介导的新重复基因的起源和进化研究(Nature Ecology & Evolution 2022、Nature Communications 2021)。两个团队的合作推动了对 TnpB 的挖掘。该研究得到了科技部、中国科学院先导专项、农业部和国家自然科学基金委等项目的资助。
Nature Biotechnology 杂志同时发表了 Research Briefing 文章对该研究成果进行了总结和展望:Hypercompact genome editors are discovered by mining a transposon family。之外,应 Nature Portfolio Communities 的邀请,研究团队在 “Behind the Paper” 栏目撰稿分享了该项工作背后的故事,详细内容请见期刊官网。
相关人物
















