AlphaFold 3来了
 发布时间:2024-05-10
发布时间:2024-05-10
 信息来源:中国科学报
字体:
大
中
小
信息来源:中国科学报
字体:
大
中
小
 打印
打印
 发布时间:2024-05-10
发布时间:2024-05-10
 信息来源:中国科学报
信息来源:中国科学报
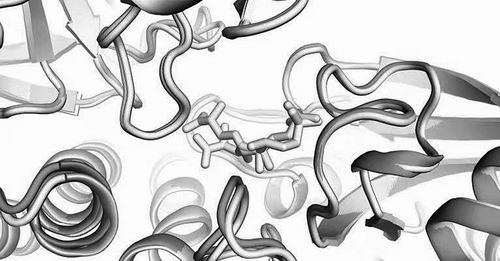
与化学物质结合的细菌酶的AlphaFold 3模型。图片来源:Isomorphic实验室
5月8日出版的《自然》报道了AlphaFold 3能以较高准确率预测蛋白质与其他生物分子相互作用的结构。AlphaFold 3是该人工智能模型的最新迭代,准确率与之前的专用工具相比显著提升,该模型由谷歌DeepMind和Isomorphic实验室的团队研发。
这一最新模型能够预测蛋白质数据银行(Protein Data Bank)内几乎所有分子类型复合物的结构。这种用计算机解析蛋白质与其他分子复杂相互作用的能力,将增进研究人员对生物过程的理解,并有望推动药物研发。
AlphaFold于2020年问世,它和迭代版AlphaFold 2能够根据蛋白质的氨基酸(蛋白质的基本成分)序列预测其3D结构。之后的AlphaFold-Multimer推动了对蛋白质-蛋白质复合物的预测。不过,由于不同类型蛋白质的相互作用差异太大,因此用单一深度学习模型预测复合物的范围一直很难。
在新研究中,DeepMind的John Jumper和同事报告说,在AlphaFold 2模型的深度学习架构和训练系统得到大幅提升的情况下,如今AlphaFold 3可以对一个统一框架内大量生物分子系统的结构进行更准确的预测。
AlphaFold 3能预测蛋白质与其他蛋白质、核酸、小分子、离子、修饰蛋白质残基的复合物,以及抗体-抗原相互作用。预测准确性显著超过当前预测工具。
同时,研究人员也指出了一些局限性,比如约4.4%的结构会出现不正确的手性(一种对称特性)。他们补充说,进一步提升模拟的准确率需要生成一个很大的预测集并对预测结构进行排序,而这会产生额外的计算成本。(记者 赵熙熙)
相关论文信息:
https://doi.org/10.1038/s41586-024-07487-w
相关人物
















